Microbial Genetic Algorithm
学习资料:
要点 ¶
如果对遗传算法有兴趣的朋友, 强烈推荐先看看我制作的动画短片 什么是遗传算法, 在动画里有了基础的了解, 在接下来的内容中, 你就如鱼得水啦.
说到遗传算法 (GA), 有一点不得不提的是如何有效保留好的父母 (Elitism), 让好的父母不会消失掉. 这也是永远都给自己留条后路的意思. Microbial GA (后面统称 MGA) 就是一个很好的保留 Elitism 的算法. 一句话来概括: 在袋子里抽两个球, 对比两个球, 把球大的放回袋子里, 把球小的变一下再放回袋子里, 这样在这次选着中, 大球不会被改变任何东西, 就被放回了袋子, 当作下一代的一部分.
算法 ¶
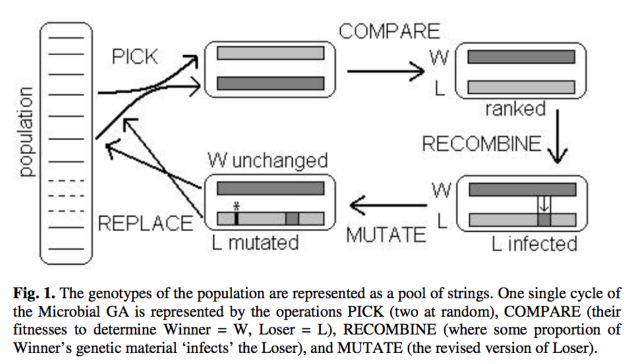
像最开始说的那样, 我们有一些 population, 每次在进化的时候, 我们会从这个 pop 中随机抽 2 个 DNA 出来,
然后对比一下他们的 fitness, 我们将 fitness 高的定义成 winner, 反之是 loser. 我们不会去动任何 winner 的 DNA,
要动手脚的只有这个 loser, 比如对 loser 进行 crossover 和 mutate. 动完手脚后将 winner 和 loser 一同放回 pop 中.
通过这样的流程, 我们就不用担心有时候变异着变异着, 那些原本好的 pop 流失掉了, 有了这个 MGA 算法, winner 总是会被保留下来的.
GA 中的 Elitism 问题通过这种方法巧妙解决了.
进化啦 ¶
这次我们还是通过之前那个找曲线中最大点的方式诠释 MGA 算法. class 中的结构框架基本没变, 只是少了 select 的功能.
因为我们会将 select 功能写在 evolve 中. 这样方便点.
class MGA:
def crossover(self, loser_winner):
def mutate(self, loser_winner):
def evolve(self, n):
首先我们先说 evolve, 在这个功能中, 两只手进入袋子去抓两个 DNA 出来的动作要进行 n 次, 然后每一次评估一下这两个抓出来的 DNA 的 fitness 怎么样.
这样我们就能定义这两个中, 哪个是 winner, 哪个是 loser. 对于 loser, 我们将 winner 的一部分 DNA crossover 去 loser 那, 期望 loser 有了 winner 的这一部分基因能变好一点.
然后 loser 再通过一部分几率 mutate 一下. 所以在 evolve 中的算法就是这样写:
class MGA:
def evolve(self, n): # nature selection wrt pop's fitness
for _ in range(n): # random pick and compare n times
sub_pop_idx = np.random.choice(np.arange(0, self.pop_size), size=2, replace=False)
sub_pop = self.pop[sub_pop_idx] # pick 2 from pop
product = F(self.translateDNA(sub_pop))
fitness = self.get_fitness(product)
loser_winner_idx = np.argsort(fitness)
loser_winner = sub_pop[loser_winner_idx] # the first is loser and second is winner
loser_winner = self.crossover(loser_winner)
loser_winner = self.mutate(loser_winner)
self.pop[sub_pop_idx] = loser_winner
crossover 和 mutate 都是按上面的说法, 只在 winner_loser 中进行. 因为这里的 DNA 是用二进制编码的. 如果不明白这里发生了什么,
请看到这个教程里面的详细说明.
class MGA:
def crossover(self, loser_winner): # crossover for loser
cross_idx = np.empty((self.DNA_size,)).astype(np.bool)
for i in range(self.DNA_size):
cross_idx[i] = True if np.random.rand() < self.cross_rate else False # crossover index
loser_winner[0, cross_idx] = loser_winner[1, cross_idx] # assign winners genes to loser
return loser_winner
def mutate(self, loser_winner): # mutation for loser
mutation_idx = np.empty((self.DNA_size,)).astype(np.bool)
for i in range(self.DNA_size):
mutation_idx[i] = True if np.random.rand() < self.mutate_rate else False # mutation index
# flip values in mutation points
loser_winner[0, mutation_idx] = ~loser_winner[0, mutation_idx].astype(np.bool)
return loser_winner
如果觉得看整体代码可能方便理解的话, 请去往我的 github 中查看整套代码. 最后套上训练的循环, 就完事啦.
ga = MGA(...)
for generation in range(N_GENERATIONS):
ga.evolve(n=5)
文章里面的代码都是简化版的, 如果要看到完整版和可视化的代码, 请去往我的 github.
分享到:
![]()
![]()
![]()
![]()
如果你觉得这篇文章或视频对你的学习很有帮助, 请你也分享它, 让它能再次帮助到更多的需要学习的人.
莫烦没有正式的经济来源, 如果你也想支持 莫烦Python 并看到更好的教学内容, 赞助他一点点, 作为鼓励他继续开源的动力.