为什么用 Numpy 还是慢, 你用对了吗?
这原本是我写在知乎上的一篇文章, 觉得还是转到这个网站上作为一个教程比较好
最近在写代码, 编一个 Python 模拟器, 做 simulation, 好不容易用传说中 Python 里速度最快的计算模块 “Numpy” 的写好了, 结果运行起来, 出奇的慢! 因为一次simulation要一个小时, 要不停测试, 所以自己受不了了.. 首先, 我的脑海中的问题, 渐渐浮现出来.
- 我知道 Pandas 要比 Numpy 慢, 所以我尽量避免用 Pandas. 但是 Numpy (速度怪兽), 为什么还是这么慢?
带有写代码洁癖的我好好给 google 了一番. 第一个出现在我眼前的就是这个文章, Getting the Best Performance out of NumPy. 所以我也将自己从这个文章中学到的诀窍分享给大家, 并补充一些内容.
为什么用 Numpy? ¶
我们都知道, Python 是慢的, 简单来说, 因为 Python 执行你代码的时候会执行很多复杂的 “check” 功能, 比如当你赋值
b=1; a=b/0.5
这个运算看似简单, 但是在计算机内部, b 首先要从一个整数 integer 转换成浮点数 float, 才能进行后面的 b/0.5, 因为得到的要是一个小数. 还有很多其他的原因和详细说明 (比如 Python 对内存的调用) 在这里能够找到:
Why Python is Slow: Looking Under the Hood
提到 Numpy, 它就是一个 Python 的救星. 能把简单好用的 Python 和高性能的 C 语言合并在一起. 当你调用 Numpy 功能的时候, 他其实调用了很多 C 语言而不是纯 Python. 这就是为什么大家都爱用 Numpy 的原因.
创建 Numpy Array 的结构 ¶
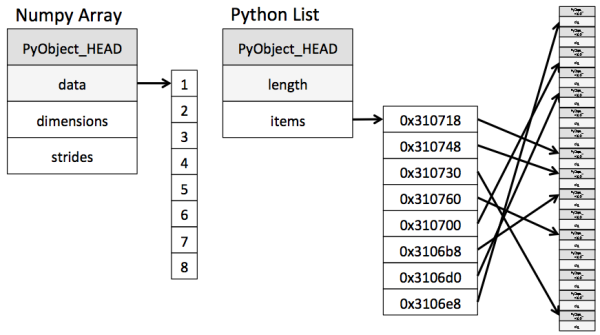
其实 Numpy 就是 C 的逻辑, 创建存储容器 “Array” 的时候是寻找内存上的一连串区域来存放, 而 Python 存放的时候则是不连续的区域, 这使得 Python 在索引这个容器里的数据时不是那么有效率. Numpy 只需要再这块固定的连续区域前后走走就能不费吹灰之力拿到数据. 下图是来自 Why Python is Slow: Looking Under the Hood, 他很好的解释了这一切.
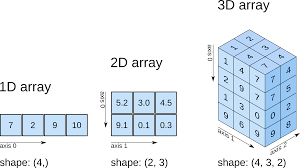
在运用 Numpy 的时候, 我们通常不是用一个一维 Array 来存放数据, 而是用二维或者三维的块来存放 (说出了学机器学习的朋友们的心声~).
因为 Numpy 快速的矩阵相乘运算, 能将乘法运算分配到计算机中的多个核, 让运算并行. 这年头, 我们什么都想 多线程/ 多进程 (再次说出了机器学习同学们的心声~). 这也是 Numpy 为什么受人喜欢的一个原因. 这种并行运算大大加速了运算速度.
那么对于这种天天要用到的2D/3D Array, 我们通常都不会想着他是怎么来的. 因为按照我们正常人的想法, 这矩阵就是矩阵, 没什么深度的东西呀. 不过这可不然! 要不然我也不会写这篇分享了. 重点来了, 不管是1D/2D/3D 的 Array, 从根本上, 它都是一个 1D array!
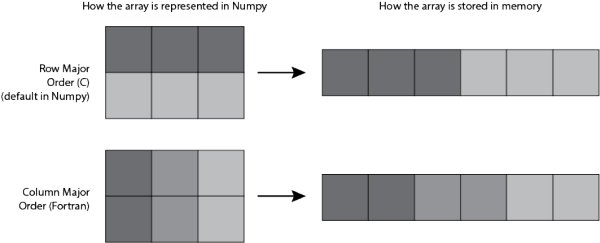
这篇 Blog的图显示. 在我们看来的 2D Array, 如果追溯到计算机内存里, 它其实是储存在一个连续空间上的. 而对于这个连续空间, 我们如果创建 Array 的方式不同, 在这个连续空间上的排列顺序也有不同. 这将影响之后所有的事情! 我们后面会用 Python 进行运算时间测试.
在 Numpy 中, 创建 2D Array 的默认方式是 “C-type” 以 row 为主在内存中排列, 而如果是 “Fortran” 的方式创建的, 就是以 column 为主在内存中排列.
col_major = np.zeros((10,10), order='C') # C-type
row_major = np.zeros((10,10), order='F') # Fortran
在 Axis 上的动作 ¶
当你的计算中涉及合并矩阵, 不同形式的矩阵创建方式会给你不同的时间效果. 因为在 Numpy 中的矩阵合并等, 都是发生在一维空间里, ! 不是我们想象的二维空间中!
a = np.zeros((200, 200), order='C')
b = np.zeros((200, 200), order='F')
N = 9999
def f1(a):
for _ in range(N):
np.concatenate((a, a), axis=0)
def f2(b):
for _ in range(N):
np.concatenate((b, b), axis=0)
t0 = time.time()
f1(a)
t1 = time.time()
f2(b)
t2 = time.time()
print((t1-t0)/N) # 0.000040
print((t2-t1)/N) # 0.000070
从上面的那张图, 可以想到, row 为主的存储方式, 如果在 row 的方向上合并矩阵, 将会更快. 因为只要我们将思维放在 1D array 那,
直接再加一个 row 放在1D array 后面就好了, 所以在上面的测试中, f1 速度要更快. 但是在以 column 为主的系统中,
往 1D array 后面加 row 的规则变复杂了, 消耗的时间也变长. 如果以 axis=1 的方式合并, “F” 方式的 f2 将会比 “C” 方式的 f1 更好.
还有一个要提的事情, 为了图方便, 有时候我会直接使用 np.stack 来代替 np.concatenate,
因为这样可以少写一点代码, 不过使用上面的形式, 通过上面的测试发现是这样. 所以之后为了速度, 我推荐还是尽量使用 np.concatenate.
np.vstack((a,a)) # 0.000063
np.concatenate((a,a), axis=0) # 0.000040
或者有时候在某个 axis 上进行操作, 比如对上面用 “C-type” 创建的 a 矩阵选点:
indices = np.random.randint(0, 100, size=10, dtype=np.int32)
a[indices, :] # 0.000003
a[:, indices] # 0.000006
因为 a 是用 row 为主的形式储存, 所以在 row 上面选数据要比在 column 上选快很多! 对于其他的 axis 的操作, 结果也类似. 所以你现在懂了吧, 看自己要在哪个 axis 上动的手脚多, 然后再创建合适于自己的矩阵形式 (“C-type”/”Fortran”).
copy慢 view快 ¶
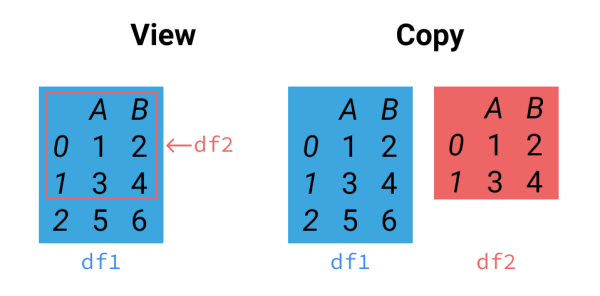
在 Numpy 中, 有两个很重要的概念, copy 和 view. copy 顾名思义, 会将数据 copy 出来存放在内存中另一个地方, 而 view 则是不 copy 数据, 直接取源数据的索引部分. 下图来自 Understanding SettingwithCopyWarning in pandas
上面说的是什么意思呢? 我们直接看代码.
a = np.arange(1, 7).reshape((3,2))
a_view = a[:2]
a_copy = a[:2].copy()
a_copy[1,1] = 0
print(a)
"""
[[1 2]
[3 4]
[5 6]]
"""
a_view[1,1] = 0
print(a)
"""
[[1 2]
[3 0]
[5 6]]
"""
简单说, a_view 的东西全部都是 a 的东西, 动 a_view 的任何地方, a 都会被动到, 因为他们在内存中的位置是一模一样的, 本质上就是自己.
而 a_copy 则是将 a copy 了一份, 然后把 a_copy 放在内存中的另外的地方, 这样改变 a_copy, a 是不会被改变的.
那为什么要提这点呢? 因为 view 不会复制东西, 速度快! 我们来测试一下速度.
下面的例子中 a*=2 就是将这个 view 给赋值了, 和 a[:] *= 2 一个意思, 从头到尾没有创建新的东西. 而 b = 2*b 中, 我们将 b 赋值给另外一个新建的 b.
a = np.zeros((1000, 1000))
b = np.zeros((1000, 1000))
N = 9999
def f1(a):
for _ in range(N):
a *= 2 # same as a[:] *= 2
def f2(b):
for _ in range(N):
b = 2*b
print('%f' % ((t1-t0)/N)) # f1: 0.000837
print('%f' % ((t2-t1)/N)) # f2: 0.001346
对于 view 还有一点要提, 你是不是偶尔有时候要把一个矩阵展平, 用到 np.flatten() 或者 np.ravel(). 他俩是不同的! ravel 返回的是一个 view (谢谢知乎上评论的提醒, 官方说如果用 ravel, 需要 copy 的时候才会被 copy , 我想这个时候可能是把 ravel 里面 order 转换的时候, 如 ‘C-type’ -> ‘Fortran’), 而 flatten 返回的总是一个 copy. 现在你知道谁在拖你的后腿了吧! 下面的测试证明, 相比于 flatten, ravel 是神速.
def f1(a):
for _ in range(N):
a.flatten()
def f2(b):
for _ in range(N):
b.ravel()
print('%f' % ((t1-t0)/N)) # 0.001059
print('%f' % ((t2-t1)/N)) # 0.000000
选择数据 ¶
选择数据的时候, 我们常会用到 view 或者 copy 的形式. 我们知道了, 如果能用到 view 的, 我们就尽量用 view, 避免 copy 数据. 那什么时候会是 view 呢? 下面举例的都是 view 的方式:
a_view1 = a[1:2, 3:6] # 切片 slice
a_view2 = a[:100] # 同上
a_view3 = a[::2] # 跳步
a_view4 = a.ravel() # 上面提到了
... # 我只能想到这些, 如果还有请大家在评论里提出
那哪些操作我们又会变成 copy 呢?
a_copy1 = a[[1,4,6], [2,4,6]] # 用 index 选
a_copy2 = a[[True, True], [False, True]] # 用 mask
a_copy3 = a[[1,2], :] # 虽然 1,2 的确连在一起了, 但是他们确实是 copy
a_copy4 = a[a[1,:] != 0, :] # fancy indexing
a_copy5 = a[np.isnan(a), :] # fancy indexing
... # 我只能想到这些, 如果还有请大家在评论里提出
Numpy 给了我们很多很自由的方式选择数据, 这些虽然都很方便, 但是如果你可以尽量避免这些操作, 你的速度可以飞起来.
在上面提到的 blog 里面, 他提到了, 如果你还是喜欢这种 fancy indexing 的形式, 我们也是可以对它加点速的. 那个 blog 中指出了两种方法
1.使用 np.take(), 替代用 index 选数据的方法.
上面提到了如果用index 来选数据, 像 a_copy1 = a[[1,4,6], [2,4,6]], 用 take 在大部分情况中会比这样的 a_copy1 要快.
a = np.random.rand(1000000, 10)
N = 99
indices = np.random.randint(0, 1000000, size=10000)
def f1(a):
for _ in range(N):
_ = np.take(a, indices, axis=0)
def f2(b):
for _ in range(N):
_ = b[indices]
print('%f' % ((t1-t0)/N)) # 0.000393
print('%f' % ((t2-t1)/N)) # 0.000569
2.使用 np.compress(), 替代用 mask 选数据的方法.
上面的 a_copy2 = a[[True, True], [False, True]] 这种就是用 TRUE, FALSE 来选择数据的. 测试如下:
mask = a[:, 0] < 0.5
def f1(a):
for _ in range(N):
_ = np.compress(mask, a, axis=0)
def f2(b):
for _ in range(N):
_ = b[mask]
print('%f' % ((t1-t0)/N)) # 0.028109
print('%f' % ((t2-t1)/N)) # 0.031013
非常有用的 out 参数 ¶
不深入了解 numpy 的朋友, 应该会直接忽略很多功能中的这个 out 参数 (之前我从来没用过). 不过当我深入了解了以后, 发现他非常有用! 比如下面两个其实在功能上是没差的, 不过运算时间上有差, 我觉得可能是 a=a+1 要先转换成 np.add() 这种形式再运算, 所以前者要用更久一点的时间.
a = a + 1 # 0.035230
a = np.add(a, 1) # 0.032738
如果是上面那样, 我们就会触发之前提到的 copy 原则, 这两个被赋值的 a, 都是原来 a 的一个 copy, 并不是 a 的 view. 但是在功能里面有一个 out 参数, 让我们不必要重新创建一个 a. 所以下面两个是一样的功能, 都不会创建另一个 copy. 不过可能是上面提到的那个原因, 这里的运算时间也有差.
a += 1 # 0.011219
np.add(a, 1, out=a) # 0.008843
带有 out 的 numpy 功能都在这里:Universal functions. 所以只要是已经存在了一个 placeholder (比如 a), 我们就没有必要去再创建一个, 用 out 方便又有效.
给数据一个名字 ¶
我喜欢用 pandas, 因为 pandas 能让你给数据命名, 用名字来做 index. 在数据类型很多的时候, 名字总是比 index 好记太多了, 也好用太多了. 但是 pandas 的确比 numpy 慢. 好在我们还是有途径可以实现用名字来索引. 这就是 structured array. 下面 a/b 的结构是一样的, 只是一个是 numpy 一个是 pandas.
a = np.zeros(3, dtype=[('foo', np.int32), ('bar', np.float16)])
b = pd.DataFrame(np.zeros((3, 2), dtype=np.int32), columns=['foo', 'bar'])
b['bar'] = b['bar'].astype(np.float16)
"""
# a
array([(0, 0.), (0, 0.), (0, 0.)],
dtype=[('foo', '<i4'), ('bar', '<f2')])
# b
foo bar
0 0 0.0
1 0 0.0
2 0 0.0
"""
def f1(a):
for _ in range(N):
a['bar'] *= a['foo']
def f2(b):
for _ in range(N):
b['bar'] *= b['foo']
print('%f' % ((t1-t0)/N)) # 0.000003
print('%f' % ((t2-t1)/N)) # 0.000508
可以看出来, numpy 明显比 pandas 快很多. 如果需要使用到不同数据形式, numpy 也是可以胜任的, 并且在还保持了快速的计算速度. 至于 pandas 为什么比 numpy 慢, 因为 pandas data 里面还有很多七七八八的数据, 记录着这个 data 的种种其他的特征. 这里还有更全面的对比: Numpy Vs Pandas Performance Comparison
如果大家还有其他的小技巧或者是速度大比拼, 欢迎在下面讨论. (一切为了速度~)
最后, 如果你对机器学习感兴趣, 这里有很多厉害的短片形式机器学习方法介绍和很多机器学习的 Python 实践教程, 让你可以用业余时间秒懂机器学习.
分享到:
![]()
![]()
![]()
![]()
如果你觉得这篇文章或视频对你的学习很有帮助, 请你也分享它, 让它能再次帮助到更多的需要学习的人.
莫烦没有正式的经济来源, 如果你也想支持 莫烦Python 并看到更好的教学内容, 赞助他一点点, 作为鼓励他继续开源的动力.